Mikrobiologische Datenbank der FU Berlin
Die Freie Universität Berlin benötigte eine mikrobiologische Primärdatenbank, um im Rahmen von Forschung und Diagnostik Bakterien-Stämme in großen Mengen mit deren diversen Eigenschaften (Standort der Probe, Verwahrart, Eigenschaften des Stammes, verwendete Methoden, etc.) verwalten zu können.
In von uns geführten Workshops mit Vertretern der Zielgruppen der Software wurden die verschiedenen Anforderungen herausgearbeitet und anschließend umgesetzt.
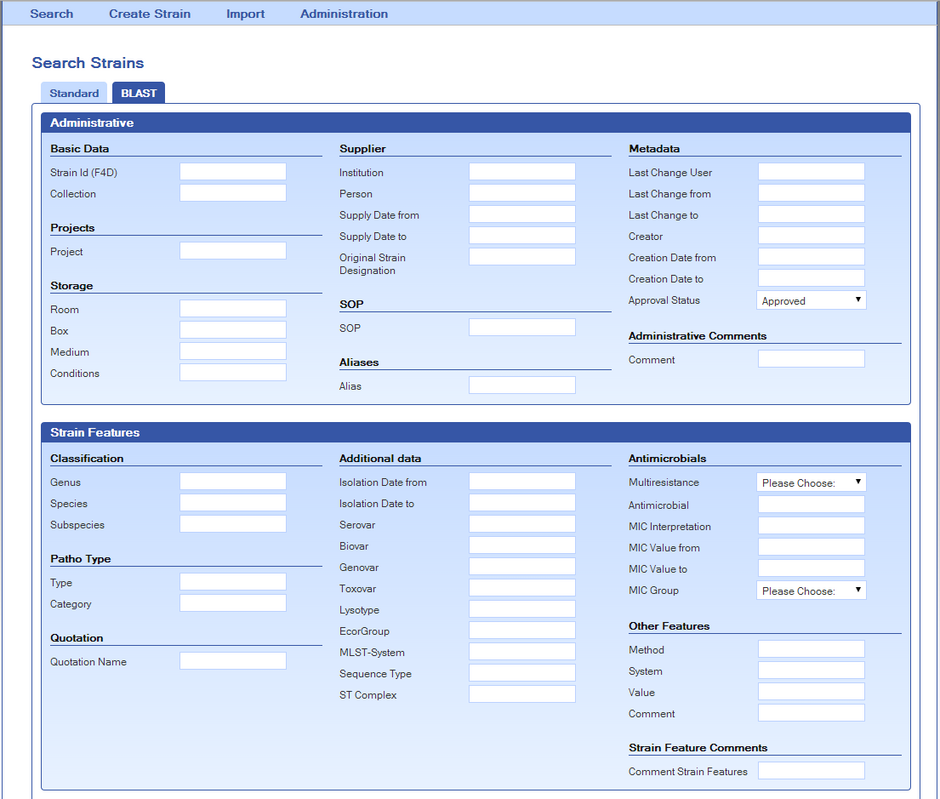
Die Anwendung sollte in einer Client-Server-Architektur umgesetzt werden. Dabei war sowohl die Suche nach jeder der Stamm-Eigenschaften sowie nach Gensequenzen mit Hilfe von BLAST von großer Bedeutung. Aber auch der Möglichkeit des Austauschs durch Export und Import gängiger Datenformate galt besonderes Augenmerk.
Mit Hilfe verschiedener Rollen ist sowohl eine rein lesende Recherche als auch eine redaktionelle Pflege oder auch eine administrative Verwaltung des Systems möglich.
Features
- Redaktionssystem mit verschiedenen Rollen/Sichtbarkeiten
- Nutzerverwaltung und Datenverwaltung
- Suche anhand aller Eigenschaften eines Stammes
- durch den Benutzer anpassbare Ausgabefelder eines Stammes
- BLAST-Suche nach Gensequenzen
- Export und Import (XLS, CSV), durch den Benutzer anpassbar
Leistungen
- Anforderungsanalyse
- Technische Konzeption
- Projektmanagement
- Technische Beratung
- Migrationskonzept und Migration
Technische Umsetzung
- Java Enterprise, Spring
- Google Web Toolkit (GWT)
- HTML, JavaScript
- PostgreSQL
- Apache Lucene
- Basic Local Alignment Search Tool (BLAST)